次世代DNAシーケンサを併用した新しいRNA関連酵素の解析法(tRNA-MaP)を開発
tRNAm1A22メチル化酵素の基質tRNA特異性を一挙に特定
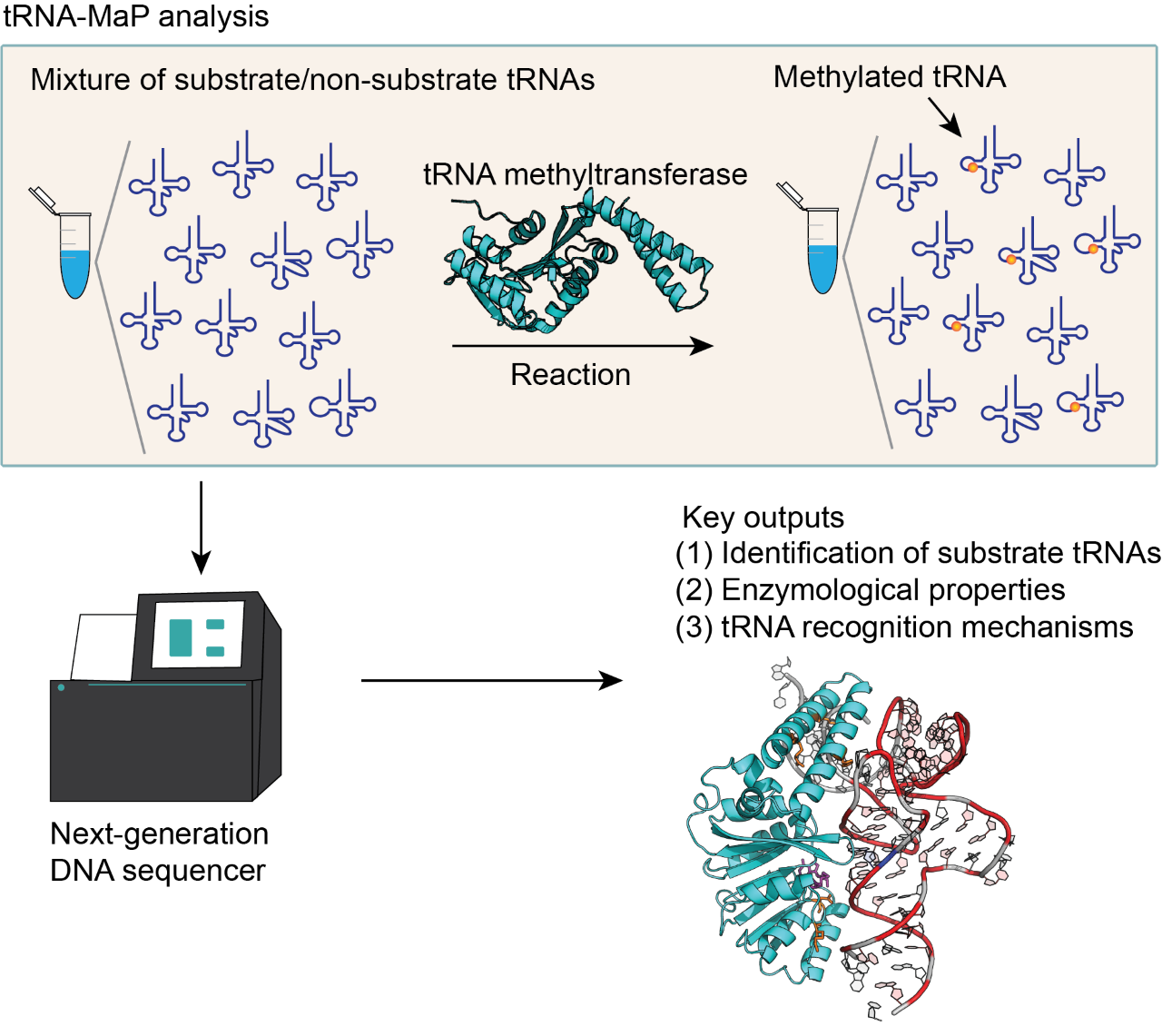
愛媛大学大学院理工学研究科の山上龍太助教と堀弘幸教授らの研究グループは、次世代DNAシーケンサを併用したtRNAメチル化酵素の新しい機能解析手法を開発し、tRNA m1A22メチル化酵素の基質tRNA特異性を一度の実験で一挙に特定する技術の開発に成功しました。本研究の成果は、米国生化学・分子生物学会が発行しているJournal of Biological Chemistry誌に2022年11月30日付けで先行公開されました。
DNA上にコードされた遺伝情報は、メッセンジャーRNAに写し取られ、タンパク質合成の際に、トランスファーRNA (tRNA)によって読み取られます。tRNAは、タンパク質合成装置であるリボソームにアミノ酸を供給する役割を担い、タンパク質合成の要として働きます。tRNAは、tRNA修飾酵素によって、部位特異的に修飾が施され、その生理的機能を獲得します。言い換えると、tRNA修飾酵素は、基質tRNAの標的部位に修飾を施すことで、tRNAの機能性獲得に貢献します。よって、「tRNA修飾酵素が、どうやって基質tRNAと非基質tRNAを選別し、さらに、いつ、どこで、どれくらいの基質tRNAを修飾するか」を理解することは、生命の普遍的な原理であるタンパク質合成機構を理解することに直結します。しかしながら、これまでの手法は非常に時間と労力がかかり、短時間でtRNA修飾酵素の特異性を網羅的に分析する手法の開発が待たれていました。
この問題を解決するために、愛媛大学大学院理工学研究科の山上龍太助教と堀弘幸教授は、次世代DNAシーケンサーを使った網羅解析手法をtRNA修飾酵素の機能解析に応用し、tRNA-MaPと呼ばれる手法を考案・開発し、世界に先駆けて報告しました。本手法は、理論上、5,000種類以上のRNAプールの中から、tRNA修飾酵素の基質となるtRNAをたった一度の実験から判別可能であり、かつ、従来法と同じ測定感度を持ちます。そのため、従来では不可能とされていた数のRNAを一挙に短時間で分析することが可能となりました。本手法とタンパク質の相同解析を組み合わせて、Geobacillus stearothermophilusのtRNAが持つ修飾ヌクレオシドを解析しました。また、tRNAの22番目のアデノシンを1-メチルアデノシンに変換する酵素(tRNA m1A22 メチル化酵素TrmK)が、どうやって基質tRNAを認識するかを明らかにしました。本手法は、理論上、どのような生物・どのようなRNAであっても応用が可能であり、高い汎用性を持ちます。本手法を他のRNA分子種やRNA関連酵素の解析に応用することで、生命の遺伝情報の流れの統合的理解に貢献するものと期待されます。
参考 URL1: https://www.jbc.org/article/S0021-9258(22)01202-9/fulltext#relatedArticles
論文情報
Application of mutational profiling: new functional analyses reveal the tRNA recognition mechanism of tRNA m1A22 methyltransferase (TrmK), Ryota Yamagami and Hiroyuki Hori, Journal of Biological Chemistry, in press. DOI:https://doi.org/10.1016/j.jbc.2022.102759
助成金等
- JSPS科研費21K20638, 22K15035,20H03211
- 愛媛大学研究助成ZI07b
問い合わせ先
氏名 : 山上 龍太・堀 弘幸
電話 : 089-927-9919・089-927-8548
E-mail : yamagami.ryota.bn@ehime-u.ac.jp, hori.hiroyuki.my@ehime-u.ac.jp
所属 : 大学院理工学研究科